圖解線粒體基因的遺傳多樣性分析
遺傳多樣性又稱基因多樣性,是生物多樣性的核心組成部分,也是物種多樣性和生態多樣性的基礎。線粒體DNA (mitochondrial DNA, mtDNA) 為母系遺傳(即細胞質遺傳),具有基因組結構簡單,且不發生重組現象,突變固定后形成的多態性位點既能反映出群體遺傳特征,用于分析種群分化和種屬關系等。
01
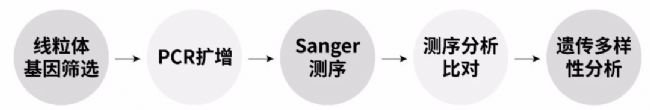
服務流程
分析內容
序列多態性分析
群體遺傳多樣性
群體單倍型分析
遺傳分化和基因流
Tajima's D和Fu's FS中性檢測
03分析方法
(方法較多,此處僅供參考)
-
BioEdit和clustalx軟件對Sanger測序結果進行分析整理;
-
MEGA 4.0軟件統計序列的堿基組成與含量、堿基信息位點數量,計算遺傳距離,并構建基于K2-P的鄰接(NJ)系統發育樹;
-
DNAsp 5.10軟件制作核苷酸岐點分布圖,統計變異位點位置,單倍型分布等;
-
Arlequin 3.1軟件精確計算Fst值,Tajima's D 和Fu's FS中性檢驗,以及進行AMOVA分析等;
-
PopART 1.7 軟件進行單倍型中介連接網絡( median-joining) 分析。
分析案例
(以COI基因檢測3個群體為例)
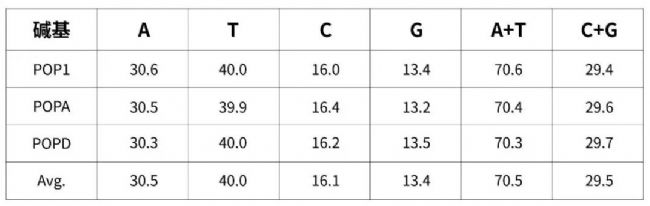
序列堿基組成:分析不同群體中基因序列的堿基組成差異。
分析結論:A+T含量(70.5%)明顯高于C+G含量(29.5% ),堿基組成具有明顯的偏向性。
表1.COI基因片段的序列組成
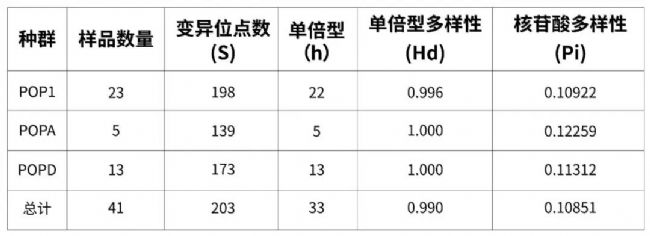
群體遺傳多樣性:通過對不同群體的基因序列突變類型進行分析,計算出群體間單倍型多樣性及核苷酸多樣性。
分析結論:共定義了33個單倍型,單倍型多樣性平均為0.990,核苷酸多樣性平均為0.10851。群體間遺傳多樣性大小差異不大。
表2.COI基因的遺傳多樣性指數
群體單倍型分析:在單倍型分析的基礎上進行分析,主要體現單倍型在各個群體的分布情況,各個單倍型間的進化關系,各個群體間的關系。
分析結論:3個群體的COI基因序列明顯聚為2個聚類簇,這3個群體間存在著一定的基因交流,并且POP1與POP2、POP3之間存在明顯的遺傳分化,POP2和POP3之間的遺傳分化較小。
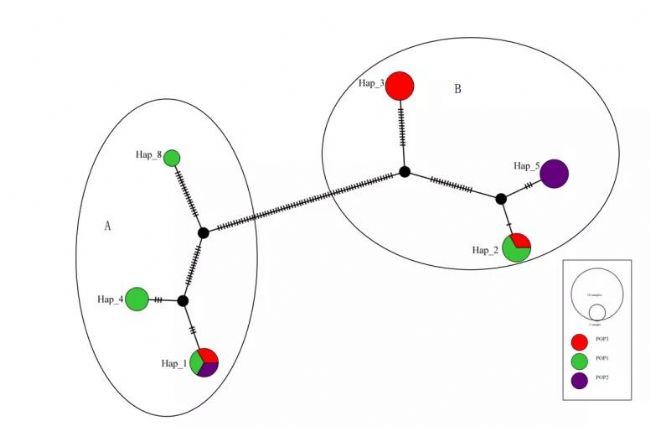
圖1.基于COI基因序列的構建單倍型網絡中介圖
NJ系統發育樹:基于遺傳距離對各個樣品進行聚類分析。
分析結論:這說明3個群體間存在著一定的基因交流,并且3個群體之間存在明顯的遺傳分化,POP2和POP3之間的遺傳分化較小。與單倍型網絡中介圖相符。
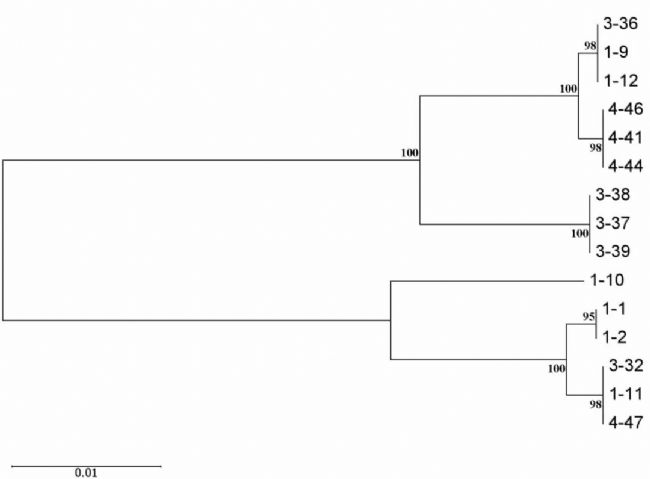
圖2.基于COI基因序列構建的NJ發育樹
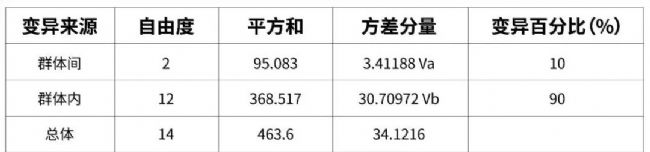
遺傳分化系數、基因流、分析方差分析:主要是看群體間基因流動情況,以及遺傳變異的主要來源。
分析結論:從群體間的遺傳分化結果顯示POP3與POP2、POP1間的基因交流頻繁。AMOVA的分析結果顯示,群體的遺傳變異主要來來自于群體內。
表3.基于COI基因的群體間Fst (對角線下)和Nm值(對角線上)
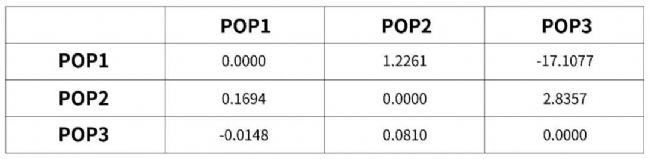
表4.種群AMOVA分析
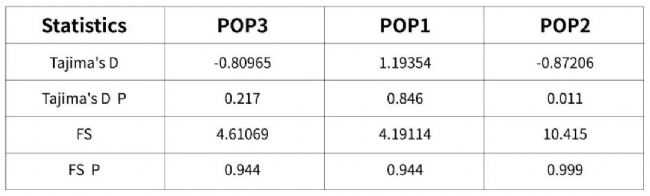
Tajima's D和Fu's FS中性檢測:判斷種群在進化歷史上是否發生過種群擴張。
分析結論:POP3和POP2個群體Tajima'sD為負值,而Fu's中性檢測值不為負值,POP1群體Tajima'sD和Fu's中性檢測值都不為負值,說明這3個地理群體可能都沒有經歷快速種群擴張。
表5.3個群體的Tajima'sD和Fu'sFS中性檢測值
以上來自擎科項目分析模板,無實際研究意義。
擎科現可提供測序+分析全套服務,歡迎咨詢!