徠卡精準空間生物學解決方案簡介(五)
Aivia 基于人工智能的圖像分析軟件
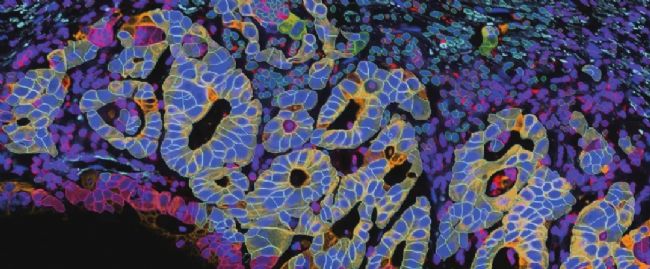
30個Biomarker標記的結腸腺癌組織切片,通過Cell DIVE系統進行成像,使用Aivia的多重細胞檢測方案和自動聚類工具進行分析。
Aivia 采用先進的基于人工智能的軟件架構,構建了一個二維至五維的圖像可視化、分析與數據詮釋的完整平臺,能夠在短短幾分鐘內可靠地處理和重建高度復雜的圖像。分析的主觀性和不易重復性是生物圖像分析中需要克服的關鍵障礙。標準分割方法會導致不符合標準的結果,因此需要進行大量的人工干預,而這很容易出錯。Aivia改變了這一切,Aivia13賦能研究者挖掘空間組學洞見。
►Aivia工作流程
原始數據的打開。
可以打開并查看
近百通道超多標圖像。
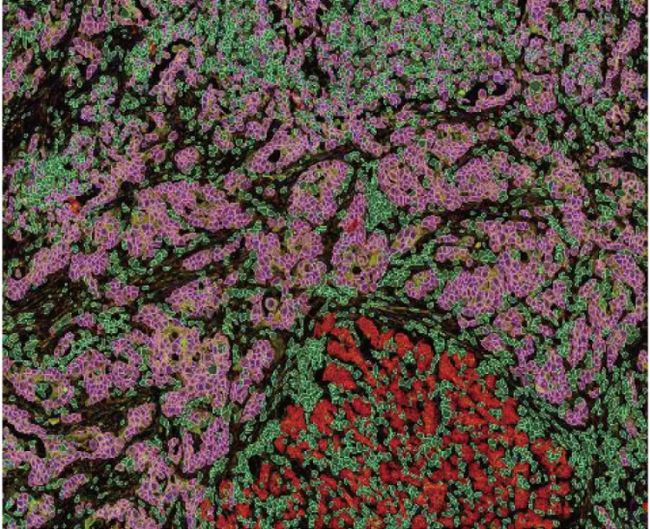
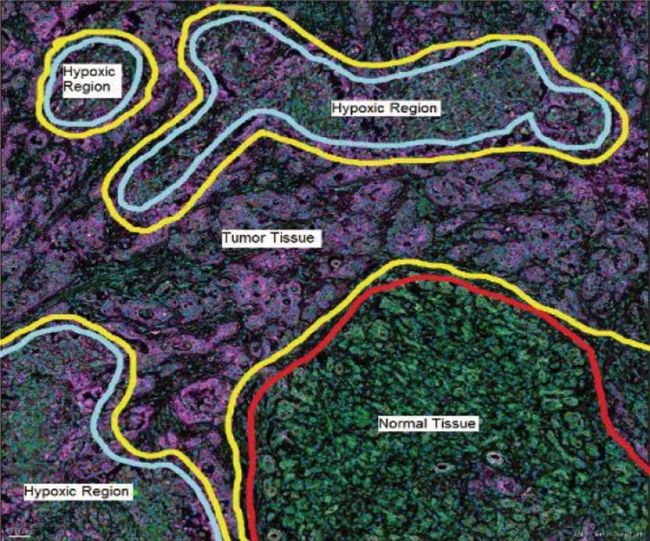
組織區域與細胞表型分布。
通過Leiden自動聚類方法進行的組織區域與細胞表型分布分析。
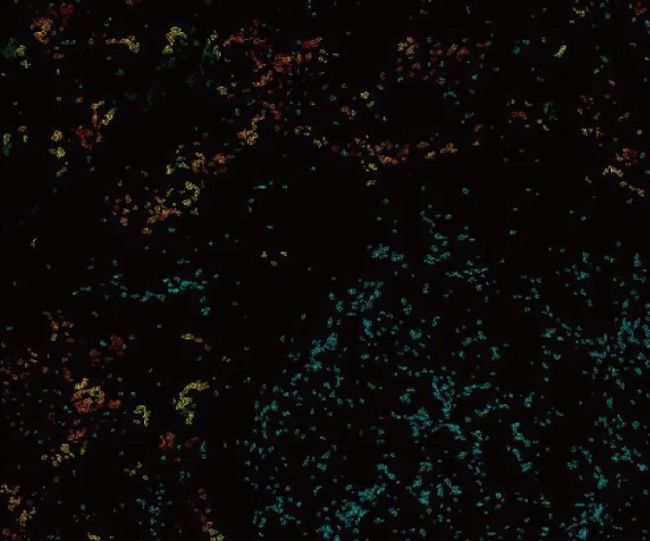
距離分析與圖像展示。高表達的GLUT1+細胞根據與免疫細胞(青色)的距離進行著色,最近的細胞顯示為紅色,最遠的顯示為綠色。
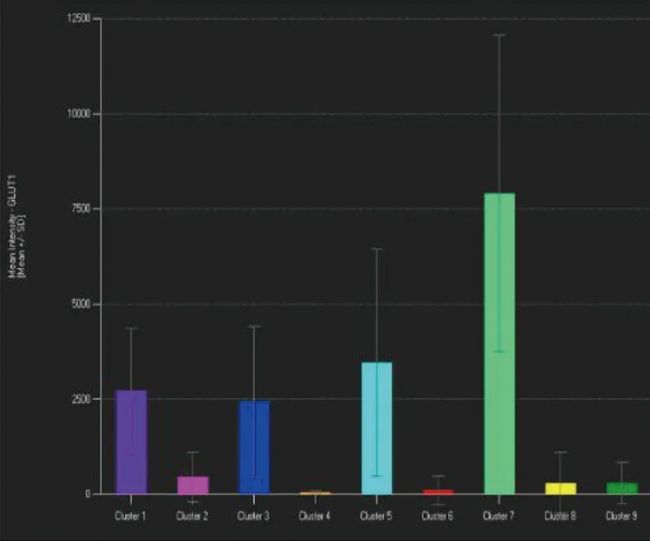
數據結果可視化⸺柱狀圖,顯示了所有聚類的一項測量的并列比較。可以選擇任何測量參數,包括標記強度或形態學。

數據結果可視化——分組散點圖。通過分組將數據點聚合到一起中,以幫助更好地查看數據。圓圈的大小對應于每個組中的數據點數量。每個組或子組以不同的顏色表示。
數據結果可視化——樹狀圖,用戶可以選擇包括標記物強度或形態在內的任何測量。樹狀圖顯示有助于跨聚類進行輕松比較。

數據結果可視化——熱圖,顯示兩項測量之間的皮爾遜相關性。
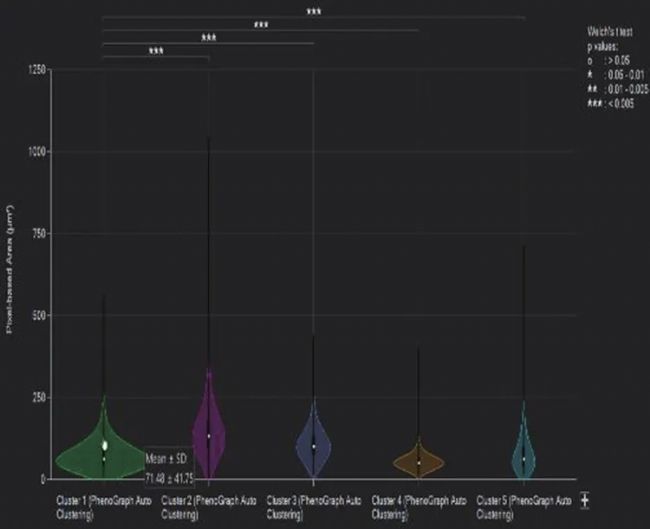
數據結果可視化——小提琴圖。展示多組數據的分布狀態以及概率密度。
降維分析是一種強大的工具,通過將數據表示為較低維度的數據,可視化和理解具有高維度的數據。Aiva中有三種維度規約方法:
1.UMAP-比t-SNE更快
2.PacMAP-比UMAP更快,并且更好地保留了高維數據的局部和全局結構
3. t-SNE
►方案特點
- 易于使用的深度學習分割和分類工具
- 打開、查看和交互大型多路復用二維圖像(最多100個通道,檢測到超過100萬個對象)
- 使用人工智能在大型多路復用二維圖像中準確分割具有不同形態學的細胞
- 分析的樣本多種多樣,包括明場、熒光,組織全景、組織ROI和組織芯片
- 使用基于人工智能的表型分析或數據驅動的無監督自動表型分析發現圖像中的細胞表型
- 使用免費的Aivia Community軟件輕松打開和交互式探索來自任何地方的2到5D顯微數據集
Aivia組織芯片識別與分析
明場分析
K-means聚類分析

DM3000傳統病理染色明場成像,通過Aivia打開數據

Pixel Classifer進行細胞圈選與分割
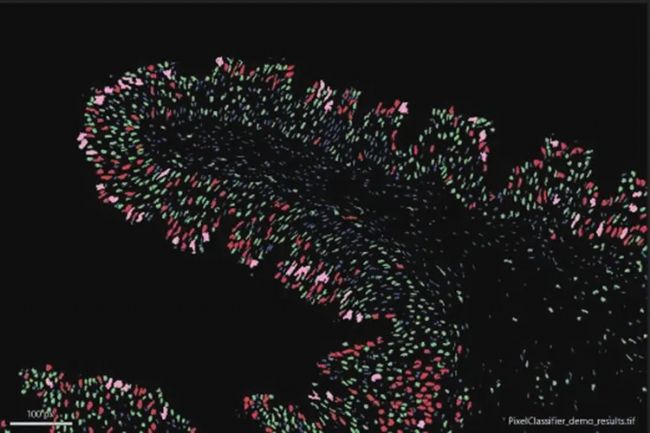
基于細胞面積進行K-means聚類分析,同一顏色代表一種細胞群體
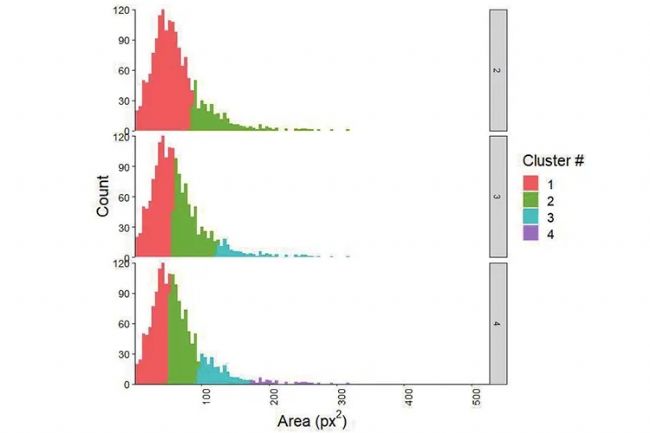
不同細胞群體按照面積大小進行數量的統計分布
熒光分析
Cell DIVE+Aivia繪制結腸腺癌免疫圖譜

Cell DIVE技術獲取結腸腺癌(CAC)組織成像。針對包括白細胞譜系、上皮細胞、基質細胞和內皮細胞類型的約30種生物標志物來表征人類結腸腺癌組織中的腫瘤免疫微環境。

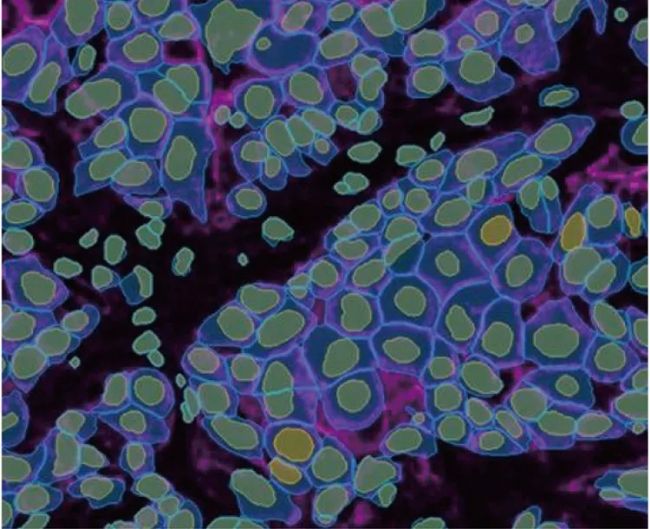
Cell DIVE原始數據(左)與Aivia 以AI為基礎的細胞膜和細胞核分割(右)
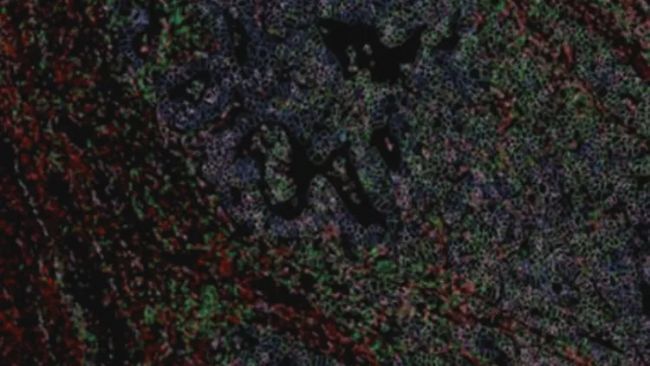
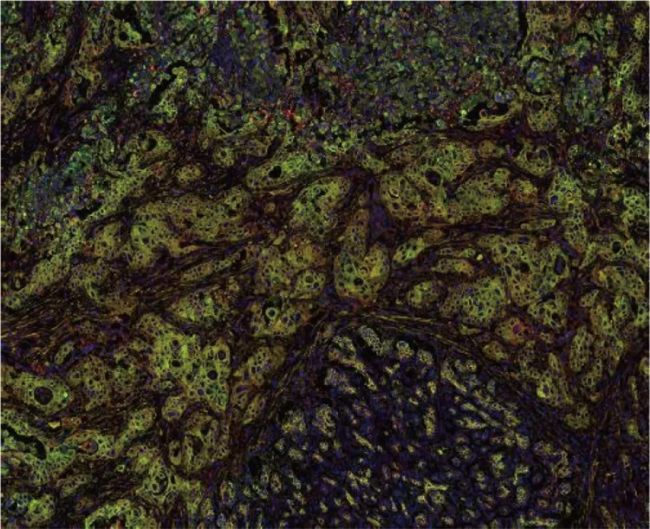
生物標記物識別與細胞分型
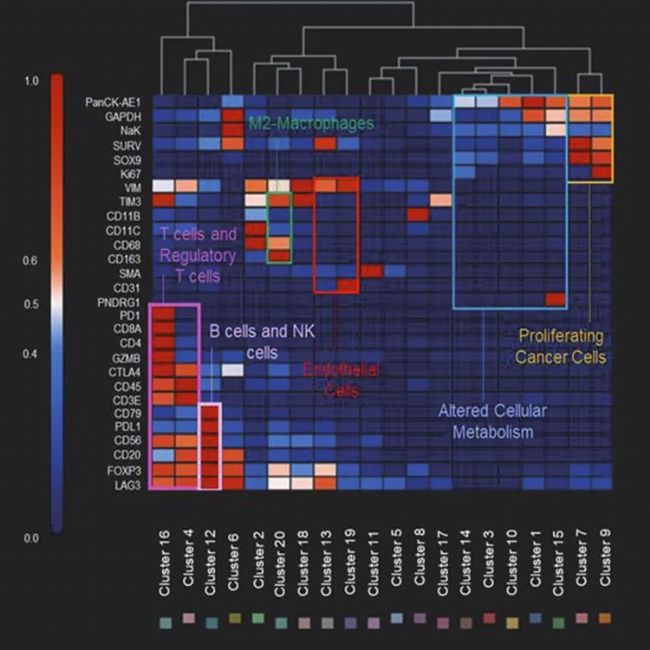
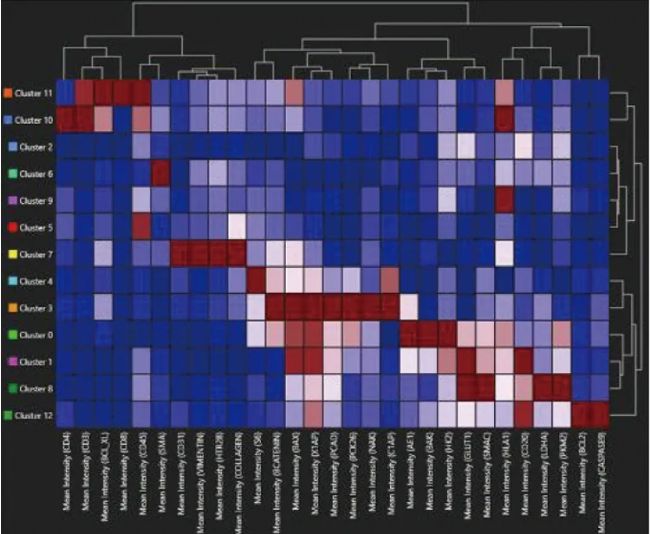
CAC內的聚類分析揭示了標記物之間的等級關系。熱圖表示給定聚類中標記物強度的測量值。使用AIVIA上的PhenoGraph Leiden算法識別的20種聚類,用來識別CAC組織中標記物之間的復雜和非線性關系
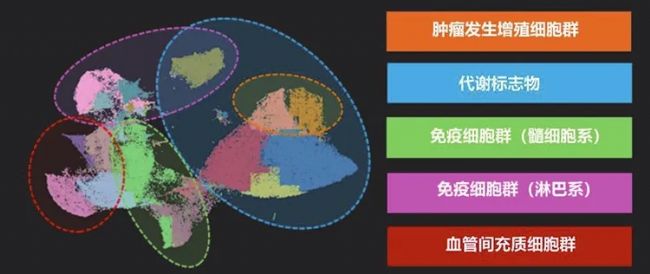
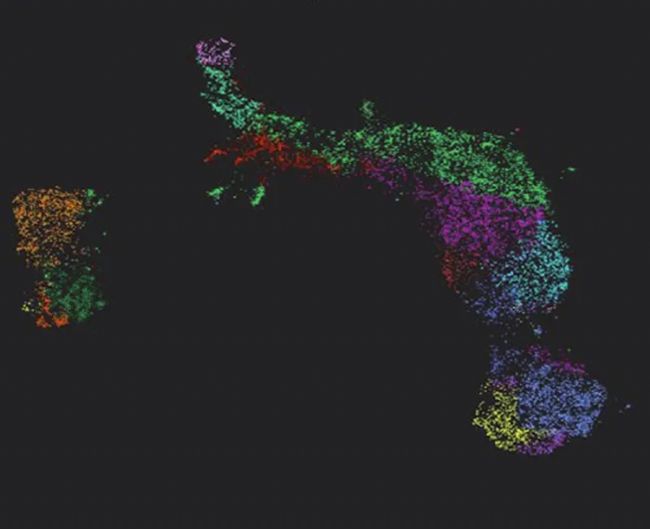
降維分析(UMAP)顯示所有已識別的表型簇,并將這些簇分組。CAC組織內的各種標志物和標志物組以腫瘤發生增殖標志物(橙色)、髓細胞標志物(綠色)、血管標志物(紅色)、淋巴標志物(粉紅色)和代謝標志物(藍色)的形式廣泛聚集
►科研成果發表(部分)
1.Stringer C, Wang T, Michaelos M, Pachitariu M. Cellpose: a generalist algorithm for cellular segmentation. Nature Methods.(2021)
2.Traag, V.A., Waltman, L. & van Eck, N.J. From Louvain to Leiden: guaranteeing well- connected communities. Sci Rep (2019).
3.MacQueen, J. Some methods for classification and analysis of multivariate observations. Proceedings of the Fifth Berkeley Symposium on Mathematical Statistics and Probability, Volume 1: Statistics.(1967).
《徠卡精準空間生物學解決方案》
了解更多:徠卡顯微